Les débuts de la GÉNÉTIQUE MOLÉCULAIRE remontent aux expériences de FRED GRIFFITH en 1928, où il infectait des souris avec différentes souches d'une bactérie pathogène, Streptococcus pneumoniae. Cette bactérie est létale pour l'homme comme pour la souris et Griffith espérait qu'en étudiant ses effets sur la souris, il pourrait mieux comprendre ses effets sur l'humain. On savait déjà qu'une mutation de la bactérie engendrant la perte de sa capsule lui faisait PERDRE SA VIRULENCE. Donc, si l'on injectait à un premier groupe de souris la bactérie capable de produire sa capsule et à un deuxième groupe celle qui en était incapable, seules les souris du premier groupe mourraient (Fig. 1). Cependant, si les bactéries encapsulées étaient tout d'abord tuées à la chaleur avant d'être injectées, les souris survivaient. Faisant l'hypothèse que malgré tout la capsule était cruciale pour la pathogénicité de la bactérie, Griffith tenta d'injecter un mélange de bactéries encapsulées mortes avec des bactéries non-encapsulées vivantes. Ce mélange tua les souris, ce qui était surprenant puisque l'injection des capsules seules n'avait aucun effet. De plus, lorsque Griffith isola les bactéries de ces souris mortes (respectant ainsi le postulat de Koch) il observa qu'elles s'étaient TRANSFORMÉES en bactéries encapsulées! Trois autres scientifiques (O.T. Avery, C.M. McLeod et M. McCarty) ont entrepris de comprendre le mécanisme de ce "PRINCIPE DE TRANSFORMATION" (PT). Ils vérifièrent que ce n'était pas le matériel capsulaire qui avait tué les souris, mais bien les bactéries vivantes qui avaient retrouvées la capacité de produire une capsule. De plus, leurs études démontrèrent que la nature génétique des bactéries encapsulées retrouvées sur les souris mortes était modifiée; ou encore que les cellules encapsulées mortes originales étaient rescucitées. La reconnaissance de ce principe de transformation, stimula de nouvelles recherches. Au départ, la plupart des scientifiques s'accordèrent à dire que le PT devait se loger au niveau des protéines, puisqu'à l'époque seules les protéines étaient reconnues comme étant assez complexes pour porter de l'information génétique. Après 10 ans de recherche, presque tous les constituants cellulaires ayant été rejetés, il a été établi que seul l'ADN pouvait être le site du PT. Une expérience décisive montra que la transformation ne pouvait pas être empêchée par l'utilisation d'enzymes digérant les protéines (des protéases) mais seulement par une enzyme nouvellement isolée, la DNase, qui dégradait l'ADN. L'annonce faite en 1944 quant à la nature chimique des gènes amorça l'ère de la génétique moléculaire.
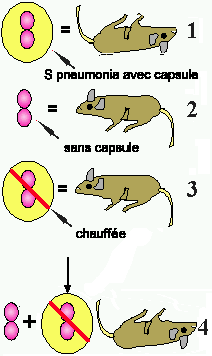
Figure 1. Première expérience de transformation.
![]() Le fonctionnement des gènes
Le fonctionnement des gènes
![]() Le transfert des gènes
Le transfert des gènes
![]() La régulation génétique
La régulation génétique
![]() Les changements ou les mutation génétiques.
Les changements ou les mutation génétiques.
RÉSUMÉ DE LA CHIMIE DES GÈNES
![]() La
structure générale des 5 bases, des pentoses et du phosphate
dans l'ADN et l'ARN peut être vue sur les sites suivants:
Cliquez
ici pour voir la structure des bases.
Cliquez
ici pour voir le pairage des bases AT & GC.
Cliquez
ici pour accéder à un résumé détaillé
de la chimie des acides nucléiques avec beaucoup d'images (programmes
utilitaires requis).
La
structure générale des 5 bases, des pentoses et du phosphate
dans l'ADN et l'ARN peut être vue sur les sites suivants:
Cliquez
ici pour voir la structure des bases.
Cliquez
ici pour voir le pairage des bases AT & GC.
Cliquez
ici pour accéder à un résumé détaillé
de la chimie des acides nucléiques avec beaucoup d'images (programmes
utilitaires requis).
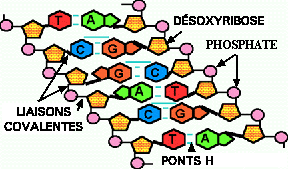
![]() L'ADN contient un phosphate, le pentose désoxyribose et les quatres
bases: adénine, thymine, guanine et cytosine.
L'ADN contient un phosphate, le pentose désoxyribose et les quatres
bases: adénine, thymine, guanine et cytosine.
![]() L'ARN contient un phosphate, le pentose ribose et les quatre bases: adénine,
uracile, guanine et cytosine.
L'ARN contient un phosphate, le pentose ribose et les quatre bases: adénine,
uracile, guanine et cytosine.
![]() Dans l'ADN double brins, les nucléotides A-T et G-C sont pairés
alors que dans l'ARN double brins les bases A-U et G-C sont pairées.
Les paires de bases sont liées par des ponts H, 2 entre A et T ou
A et U, et 3 entre G et C. Les 2 brins sont dits COMPLÉMENTAIRES.
Dans l'ADN double brins, les nucléotides A-T et G-C sont pairés
alors que dans l'ARN double brins les bases A-U et G-C sont pairées.
Les paires de bases sont liées par des ponts H, 2 entre A et T ou
A et U, et 3 entre G et C. Les 2 brins sont dits COMPLÉMENTAIRES.
![]() L'ADN est
ordinairement à
double
brins, alors que l'ARN est souvent à simple brin mais peut aussi
être doubles brins. Pour des images d'ADN, allez à la Librairie
RasMol & cherchez la section "DNA double helix".
L'ADN est
ordinairement à
double
brins, alors que l'ARN est souvent à simple brin mais peut aussi
être doubles brins. Pour des images d'ADN, allez à la Librairie
RasMol & cherchez la section "DNA double helix".
![]() L'ADN sert toujours de support au code génétique, alors que
l'ARN peut avoir d'autres fonctions que celle de transporter de l'information
génétique.
Cliquez
ici pour une comparaison de l'ADN et de l'ARN.
L'ADN sert toujours de support au code génétique, alors que
l'ARN peut avoir d'autres fonctions que celle de transporter de l'information
génétique.
Cliquez
ici pour une comparaison de l'ADN et de l'ARN.
![]() Les chaînes d'ADN et d'ARN sont composées d'une alternance
de sucres et de phosphates, avec les bases liées aux sucres.
L'image présentée
ici
montre bien comment les atomes sont liés les uns aux autres dans la molécule
d'ADN.
Les chaînes d'ADN et d'ARN sont composées d'une alternance
de sucres et de phosphates, avec les bases liées aux sucres.
L'image présentée
ici
montre bien comment les atomes sont liés les uns aux autres dans la molécule
d'ADN.
![]() En double brins, la chaîne prend la forme d'une hélice, avec
le squelette composé de sucres et de phosphates à l'extérieur
et les bases se faisant face au centre. Cette hélice est flexible
et peut être comprimée dans un petit espace.
En double brins, la chaîne prend la forme d'une hélice, avec
le squelette composé de sucres et de phosphates à l'extérieur
et les bases se faisant face au centre. Cette hélice est flexible
et peut être comprimée dans un petit espace.
Cliquez ici pour une image de la structure des chromosome et de l'ADN.
![]() Chaque longue chaîne unitaire d'ADN ou d'ARN est appelé un
CHROMOSOME
et l'ensemble du code génétique d'un organisme est le GÉNOME.
Chaque longue chaîne unitaire d'ADN ou d'ARN est appelé un
CHROMOSOME
et l'ensemble du code génétique d'un organisme est le GÉNOME.
![]() Chaque cellule-fille d'un organisme reçoit une copie complète
du génome de la cellule parentale.
Chaque cellule-fille d'un organisme reçoit une copie complète
du génome de la cellule parentale.
Les cellules procaryotes n'ont en général qu'un seul chromosome circulaire, on les dit haploïdes, alors que les eucaryotes en ont plusieurs différents et ont au moins deux copies de chaque chromosomes, ce qui en fait des diploïdes. Un procaryote comme E. coli contient approx. 4700 gènes, mais les cellules eucaryotes peuvent contenir plus de 100,000 gènes. La plus petite bactérie contient ~ 1000 gènes.
Cliquez
ici
pour une discussion plus approfondie sur la structure et la synthèse
des acides nucléiques.
Bien que la découverte de la structure 3-D de l'ADN en 1953 par Watson et Crick a permis d'expliquer la façon dont les gènes se reproduisent, les détails de ce mécanisme de réplication ne sont toujours pas pleinement compris. Lorsque l'on aborde ce phénomène, les points suivants doivent être considérés:
![]() La réplication de l'ADN procède à un train d'enfer.
Par exemple, la réplication du génome de E.coli, d'approx.
5E6 paires de bases prend toujours approximativement 40 min dans les meilleures
conditions, ce qui signifie l'addition de deux
milles paires de bases à chaque seconde. L'ADN étant
composé d'une dizaine de paires de bases par spire, ce processus
imprime un mouvement rotatif de 12000 rpm
à la molécule d'ADN!
La réplication de l'ADN procède à un train d'enfer.
Par exemple, la réplication du génome de E.coli, d'approx.
5E6 paires de bases prend toujours approximativement 40 min dans les meilleures
conditions, ce qui signifie l'addition de deux
milles paires de bases à chaque seconde. L'ADN étant
composé d'une dizaine de paires de bases par spire, ce processus
imprime un mouvement rotatif de 12000 rpm
à la molécule d'ADN!
![]() Afin d'éviter la plupart des erreurs, il doit exister des mécanismes
de révision et de correction (de
réparation) pour s'assurer que la reproduction de la
séquence de nucléotides est correcte.
Afin d'éviter la plupart des erreurs, il doit exister des mécanismes
de révision et de correction (de
réparation) pour s'assurer que la reproduction de la
séquence de nucléotides est correcte.
![]() Cependant, les erreurs de réplication, appelées
MUTATIONS,
sont des événements essentiels au processus d'évolution,
de sorte que les mécanismes de correction doivent être efficaces,
mais pas trop, pour permettre l'amélioration de l'espèce.
Cependant, les erreurs de réplication, appelées
MUTATIONS,
sont des événements essentiels au processus d'évolution,
de sorte que les mécanismes de correction doivent être efficaces,
mais pas trop, pour permettre l'amélioration de l'espèce.
![]() 1ère
étape: La
séparation des brins d'ADN afin
de permettre à chacun des brins d'être reproduit.
1ère
étape: La
séparation des brins d'ADN afin
de permettre à chacun des brins d'être reproduit.
![]() 2ème étape: Le pairage correct de
nouvelles bases sur les brins existants, afin d'assurer une
reproduction fidèle du code génétique..
2ème étape: Le pairage correct de
nouvelles bases sur les brins existants, afin d'assurer une
reproduction fidèle du code génétique..
![]() 3ème étape: la création des
liens entre les composantes de la nouvelle molécule.
Cliquez
ici pour une présentation sur la réplication de l'ADN (Voir
les figure 1 & 4). Une autre animation
ici.
3ème étape: la création des
liens entre les composantes de la nouvelle molécule.
Cliquez
ici pour une présentation sur la réplication de l'ADN (Voir
les figure 1 & 4). Une autre animation
ici.
Lors de la première étape, une enzyme, l'ADN
POLYMERASE, se lie à un site spécifique d'origine
de réplication sur l'ADN, causant la SÉPARATION
DES BRINS. Cela permet aux nucléotides (les bases A,
T, G, C) de se pairer avec leur base complémentaire sur le vieux
brin parental. Lors de la réplication, des HÉLICASES
sont responsables du déroulement de l'ADN et défont de petites
portions de l'hélice juste devant l'ADN polymérase. Puis,
une enzyme, la LIGASE, lie le squelette,
la chaîne de molécules désoxyribose-phosphate. L'ADN
polymerase a aussi la capacité de réviser la séquence
des nucléotides. Si une paire de base incorrecte est trouvée
(ex: AG, CT), l'ADN polymérase la remplace avec une paire correcte.
Cette mini-description de la transcription est loin d'être complète,
plusieurs autres enzymes et protéines étant impliquées
dans le processus, mais sera suffisante dans le cadre de ce cours.
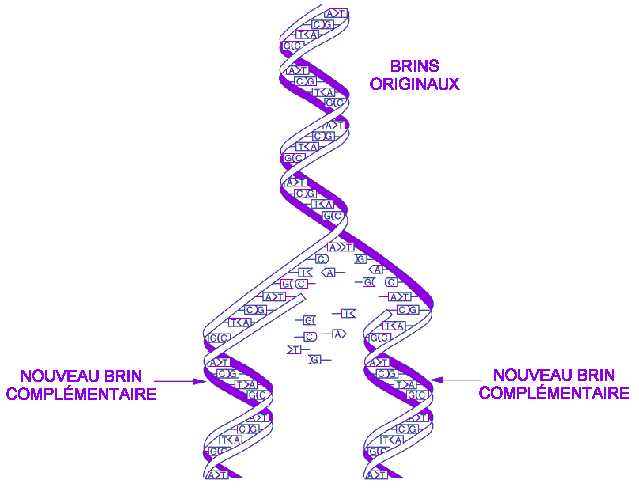
Figure 2. La réplication de l'ADN
Les études effectuées vers la fin des années 50 et le début des années 60, permirent d'élucider la relation entre le code génétique et la synthèse des protéines. Il apparut que chaque acide aminé d'une protéine devait être codé par une séquence de trois nucléotides, appelée CODON. De toute évidence, il faut qu'un codon puisse former au moins 20 "mots" génétiques différents pour coder la vingtaine d'acides aminés constituant les protéines. De plus, des mots supplémentaires doivent pouvoir donner des instructions comme "DÉBUT" et "FIN" d'une protéine. Un calcul simple montre que trois nucléotides est le minimum pour accomplir cette tâche: à deux nucléotides, on ne peut former que 4x4 = 16 mots différents, alors qu'à trois nucléotides, on forme 4x4x4 = 64 mots. Une autre pièce vint s'ajouter au casse-tête lorsqu'on repéra la présence d'une forme transitoire d'ARN à forte concentration lors de la synthèse des protéines. On posa alors l'hypothèse que cet ARN servait de messager entre l'ADN et l'appareil de synthèse des protéines, on l'appela donc ARN messager, ou ARNm. Finalement, on remarqua que la synthèse des protéines était toujours associée à une présence en masse des ribosomes, souvent liés à l'ARNm et aux protéines fraîchement produites. Ces résultats permirent d'établir une THÉORIE CENTRALE expliquant le mode de fonctionnement fondamental de tous les organismes vivants. La mise à jour de ces mécanismes de fonctionnement des unités de base de la vie révolutionnèrent notre façon de voir le monde vivant. Pour une illustration de la théorie centrale, cliquez ici.
![]() L'ARNm,
une chaîne de nucléotides, simple brin et de courte demie-vie,
transporte l'information génétique de l'ADN vers le site
de synthèse des protéines.
L'ARNm,
une chaîne de nucléotides, simple brin et de courte demie-vie,
transporte l'information génétique de l'ADN vers le site
de synthèse des protéines.
![]() Le CODON est constitué de 3
bases (nucléotides). Chaque combinaison de 3 bases code pour un
acide aminé ou un signal d'arrêt. Sur 64 codons, 61 codons
déterminent l'incorporation d'acides aminés (les
codons sens), les 3 autres sont impliqués dans la terminaison
de la traduction (codons d'arrêt, non-sens
ou stop). Le codon ATG sert à la fois à l'incorporation
de la méthionine et de codon de départ.
Le CODON est constitué de 3
bases (nucléotides). Chaque combinaison de 3 bases code pour un
acide aminé ou un signal d'arrêt. Sur 64 codons, 61 codons
déterminent l'incorporation d'acides aminés (les
codons sens), les 3 autres sont impliqués dans la terminaison
de la traduction (codons d'arrêt, non-sens
ou stop). Le codon ATG sert à la fois à l'incorporation
de la méthionine et de codon de départ.
![]() Ce code est UNIVERSEL.
Ce code est UNIVERSEL.
![]() Ce code est dégénérescent,
c'est à dire qu'il y a plusieurs codons différents pour un
même acide aminé.
Ce code est dégénérescent,
c'est à dire qu'il y a plusieurs codons différents pour un
même acide aminé.
Les différentes combinaisons de 3 nucléotides avec les acides aminés correspondants:
Deuxième position | U | C | A | G | ----+--------------+--------------+--------------+--------------+---- | UUU Phe (F) | UCU Ser (S) | UAU Tyr (Y) | UGU Cys (C) | U T U | UUC " | UCC " | UAC " | UGC " | C r P | UUA Leu (L) | UCA " | UAA STOP | UGA STOP | A o r | UUG " | UCG " | UAG STOP | UGG Trp (W) | G i e --+--------------+--------------+--------------+--------------+-- s m | CUU Leu (L) | CCU Pro (P) | CAU His (H) | CGU Arg (R) | U i i C | CUC " | CCC " | CAC " | CGC " | C è è | CUA " | CCA " | CAA Gln (Q) | CGA " | A m r | CUG " | CCG " | CAG " | CGG " | G e e --+--------------+--------------+--------------+--------------+-- | AUU Ile (I) | ACU Thr (T) | AAU Asn (N) | AGU Ser (S) | U p p A | AUC " | ACC " | AAC " | AGC " | C o o | AUA " | ACA " | AAA Lys (K) | AGA Arg (R) | A s s | AUG Met (M) | ACG " | AAG " | AGG " | G i i --+--------------+--------------+--------------+--------------+-- t t | GUU Val (V) | GCU Ala (A) | GAU Asp (D) | GGU Gly (G) | U i i G | GUC " | GCC " | GAC " | GGC " | C o o | GUA " | GCA " | GAA Glu (E) | GGA " | A n n | GUG " | GCG " | GAG " | GGG " | G ----+--------------+--------------+--------------+--------------+----note: le codon AUG, codant pour la méthionine, sert aussi de codon initiateur (Début) à la synthèse d'une protéine.
La mutation, ou changement dans la séquences des paires de bases d'un chromosome, n'est pas un phénomène extraordinaire. Bien que la fréquence de mutation soit faible (1E-9 à 1E-10 chez E. coli), le très grand nombre et la rapidité de la reproduction des cellules font en sorte que les mutations se produisent souvent. Nous-même, comme tous les organismes vivants, sommes le fruit d'une multitude de mutations. Les mutations sont la force motrice de l'évolution, basée sur la plus grande variété possible. Les mutations ont les caractéristiques suivantes:
![]() Elles
sont le résultat d'un changement dans la séquence des paires
de bases d'un génome d'ADN ou un brin d'ARN.
Elles
sont le résultat d'un changement dans la séquence des paires
de bases d'un génome d'ADN ou un brin d'ARN.
![]() La plupart des mutations ont un effet NÉFASTE,
quelques unes n'ont pas d'effet, et une infime proportion ont un effet
BÉNÉFIQUE.
La plupart des mutations ont un effet NÉFASTE,
quelques unes n'ont pas d'effet, et une infime proportion ont un effet
BÉNÉFIQUE.
![]() La plupart des mutations ont un effet néfaste parce qu'elles modifient
le code génétique, ce qui à son tour AFFECTE
LA STRUCTURE PRIMAIRE D'UNE PROTÉINE, et a pour effet
de changer la capacité de cette protéine à faire ce
à quoi elle était destinée à l'origine.
La plupart des mutations ont un effet néfaste parce qu'elles modifient
le code génétique, ce qui à son tour AFFECTE
LA STRUCTURE PRIMAIRE D'UNE PROTÉINE, et a pour effet
de changer la capacité de cette protéine à faire ce
à quoi elle était destinée à l'origine.
![]() Les mutations se produisent généralement AU
HASARD. Le taux de mutation peut cependant être augmenté
par des AGENTS MUTAGÈNES, tels
les rayons UV et X, les radiations atomiques et des agents chimiques tels
le benzène, etc.. Cependant, avec les techniques actuelles de biologie
moléculaire, il est possible d'imposer une MUTATION
DIRIGÉE, c'est à dire changer une paire de base
particulière, afin de modifier un acide aminé particulier sur une
protéine et observer l'effet que cela aura sur la fonction de cette
protéine.
Les mutations se produisent généralement AU
HASARD. Le taux de mutation peut cependant être augmenté
par des AGENTS MUTAGÈNES, tels
les rayons UV et X, les radiations atomiques et des agents chimiques tels
le benzène, etc.. Cependant, avec les techniques actuelles de biologie
moléculaire, il est possible d'imposer une MUTATION
DIRIGÉE, c'est à dire changer une paire de base
particulière, afin de modifier un acide aminé particulier sur une
protéine et observer l'effet que cela aura sur la fonction de cette
protéine.
![]() Une mutation peut affecter une seule paire de base ou un fragment important
d'un chromosome, portant sur plusieurs gènes. Par exemple, pour
une séquence de base initiale AATGGCAAT tous les changements suivants
(indiqués en rouge) sont des
mutations:
Une mutation peut affecter une seule paire de base ou un fragment important
d'un chromosome, portant sur plusieurs gènes. Par exemple, pour
une séquence de base initiale AATGGCAAT tous les changements suivants
(indiqués en rouge) sont des
mutations:
AATGCCAAT; AAAACGGTT; AATGAGCAAT; AATG.CAAT; AA.....AT (les "points" indiquent la perte de bases)
Une modification qui fait en sorte qu'un codon est remplacé par un autre codon qui code pour le MÊME acide aminé (DÉGÉNÉRESCENCE) est aussi une mutation silencieuse, même si elle n'a aucun effet apparent. Une mutation qui change un acide aminé pour un autre acide aminé très semblable mutation faux-sens peut ne pas avoir d'effet significatif sur la fonctionnalité d'une protéine et donc être neutre, mais peut aussi changer la chimie de la protéine et avoir un effet.
Les différents types de mutations sont listés ci-dessous:
![]() Forme sauvage du gène = la séquence de paires de bases originale.
Forme sauvage du gène = la séquence de paires de bases originale.
![]() Directe = passage
de la forme sauvage vers une forme mutante.
Directe = passage
de la forme sauvage vers une forme mutante.
![]() Réverse
= inverse de la mutation directe, qui entraîne un retour vers le
phénotype sauvage.
Réverse
= inverse de la mutation directe, qui entraîne un retour vers le
phénotype sauvage.
![]() Ponctuelle = n'affecte qu'une paire de base.
Ponctuelle = n'affecte qu'une paire de base.
![]() Non-sens =
conversion d'un codon sens en non-sens ou arrêt.
Non-sens =
conversion d'un codon sens en non-sens ou arrêt.
![]() Déletion = l'élimination d'une ou plusieurs paire de base.
Déletion = l'élimination d'une ou plusieurs paire de base.
![]() Décalage du cadre de lecture (frameshift) = l'insertion ou la déletion
de une ou deux paires de bases, entraînant un changement de cadre
de lecture pour tous les codons en aval.
Décalage du cadre de lecture (frameshift) = l'insertion ou la déletion
de une ou deux paires de bases, entraînant un changement de cadre
de lecture pour tous les codons en aval.
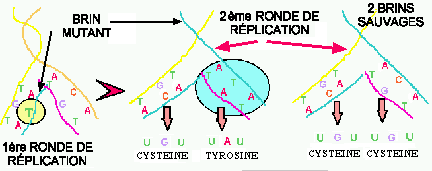
Figure 3. Fixation d'une mutation dans l'ADN. À gauche, l'ADN se réplique, cependant une erreur s'est produite (cercle jaune) et T s'est pairé avec G (brin bleu). Si cette erreur n'est pas corrigé avant la prochaine ronde de réplication, lorsque le brin contenant la paire G-T sera copiée, un des brins (image du milieu, à gauche) contiendra la sequence TGT originale et une nouvelle séquence ACA en sera tirée (bleu). Cependant, le brin de droite, qui contient la séquence incorrecte ATA sera copiée dans un nouveau brin TAT (rouge). Si la MATRICE ou BRIN INSTRUCTEUR dans cet ADN est le brin de droite dans chaque molécule d'ADN, alors l'ARNm serait copié à partir de ACA dans le brin correct pour donner un codon ARNm avec UGU pour l'acide aminé CYSTÉINE. Cependant, la MATRICE de l'ADN mutant donne un ARN mutant avec un codon UAU donnant l'acide aminé TYROSINE. Après la deuxième ronde de réplication, l'ERREUR (MUTATION) est "FIXÉE" dans chaque brin de la cellule qui contient cet ADN
Lorsqu'un gène sauvage mute, il forme une ALLÈLE ou ALTERNATIVE de ce gène. Puisque le nombre de mutations potentielles est MINIMALEMENT équivalent au nombre de paire de base dans un gène, plusieurs allèles sont possibles. La plupart des mutations résulte en un gène produisant une protéine inactive, mais dans certains cas, l'activité est améliorée ou n'est plus sujette à la régulation originale (plus de régulation, ou régulation par une molécule allostérique différente). Dans des cas très rares, l'allèle aura une activité catalytique différente, par exemple en étant moins sélective.

Figure 5. Processus de transcription.
Le processus de synthèse d'une protéine
est divisé en deux étapes. La première étape
est la
TRANSCRIPTION. Lors de ce processus,
les instructions encodées dans l'ADN sont transférées
dans l'ARN. Les raisons de la nécessité de cette étape
ne sont pas clairement établies. Ça peut être parce
que l'ARN est une molécule plus ancienne que l'ADN ou peut-être
parce que si l'ADN était utilisée pour cette étape,
il y aurait trop d'ADN dans la même cellule, rendant l'organisation
difficile. Peu importe, l'ADN est copiée dans l'ARNm. Ce processus
est mené par une enzyme, l'ARN POLYMÉRASE.
Les étapes en sont:
![]() L'ARN polymérase se positionne sur un site d'initiation, un PROMOTEUR.
Chaque gène ou groupe de gène a un promoteur. En fait, un
SITE ACTIF sur l'ARN polymérase reconnaît la séquence
du promoteur sur l'ADN comme le site de son substrat.
L'ARN polymérase se positionne sur un site d'initiation, un PROMOTEUR.
Chaque gène ou groupe de gène a un promoteur. En fait, un
SITE ACTIF sur l'ARN polymérase reconnaît la séquence
du promoteur sur l'ADN comme le site de son substrat.
![]() Une fois sur le promoteur, l'ARN polymérase ouvre l'ADN de façon
à pouvoir en copier un brin. Seul le brin instructeur ou matrice
est copié. La séquence correspondant à un gène
est situé sur seulement un des deux brins complémentaires,
mais différents gènes peuvent être encodés sur
des brins opposés.
Une fois sur le promoteur, l'ARN polymérase ouvre l'ADN de façon
à pouvoir en copier un brin. Seul le brin instructeur ou matrice
est copié. La séquence correspondant à un gène
est situé sur seulement un des deux brins complémentaires,
mais différents gènes peuvent être encodés sur
des brins opposés.
Comme on peut le voir sur la Figure 5, l'ARN polymérase descend le long de l'ADN et synthétise une copie COMPLÉMENTAIRE d'ARN à partir de la matrice.
La reproduction se poursuit jusqu'au SIGNAL D'ARRÊT,
où l'ARNm se détache du complexe ADN/ARN polymérase.
L'ARNm peut maintenant être utilisée comme une matrice par
laquelle des protéines seront produites. Ce processus de production
de protéines à partir de l'ARNm est la TRADUCTION,
dont voici les grandes lignes.
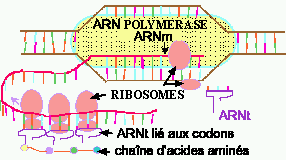
Figure 6. Traduction.
Il y a plusieurs composants impliqués dans la TRADUCTION.
Les RIBOSOMES sont des usines productrices de protéines. Le ribosome
est composé de deux sous-unités qui sont elles composées
~40 protéines et plusieurs petites molécules d'ARN. Les deux
sous-unités se joignent seulement autour de l'ARNm, sinon elles
nagent séparément dans le cytoplasme.
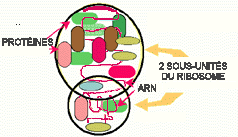
Figure 7. La structure d'un ribosome. Les autres composants sont les 20 acides aminés et une molécule appelé l'ARN de transfert ou ARNt. Le processus complet consomme beaucoup d'ATP. L'ARNm des procaryotes ne dure par longtemps étant donné que la vie des procaryotes est si courte, leur environnement peut changer rapidement et nécessiter l'arrêt d'une synthèse particulière. Il exise un ARNt unique pour chaque codon et CHAQUE ACIDE AMINÉ parce que l'ARNt doit amener le bon acide aminé au bon codon sur l'ARNm. La région sur l'ARNt qui se lie à l'ARNm est appelé l'ANTICODON.
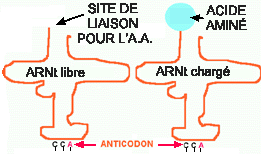
Figure 8. L'ARNt. Chaque ARNt porte un acide aminé particulier à un bout qui "matche" avec l'anticodon à l'autre extrémité. L'anticodon est complémentaire au codon sur l'ARNm.
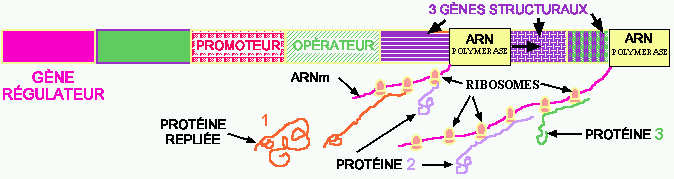
Figure 9. ARN polymérase se déplaçant le long de 3 gènes structuraux et transcriptant l'ARNm (mauve). Les ribosomes se lient aux ARNm et les traduisent en protéines (orange, vert et noir)
Les étapes de la
traduction
sont:
![]() Les deux composantes du ribosome se lient à l'ARNm au CODON
DE DÉPART (ATG, méthionine).
Les deux composantes du ribosome se lient à l'ARNm au CODON
DE DÉPART (ATG, méthionine).
![]() Puis le premier complexe ARNt-acide aminé se lie au CODON suivant
le codon de départ.
Puis le premier complexe ARNt-acide aminé se lie au CODON suivant
le codon de départ.
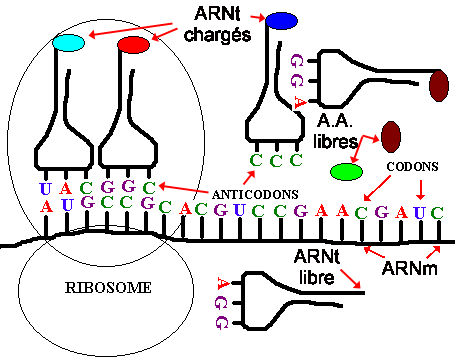
![]() L'ARNm se déplace d'un codon sur le ribosome.
L'ARNm se déplace d'un codon sur le ribosome.
![]() L'ARNt suivant, avec son acide aminé, se lie au codon libre suivant
sur l'ARNm, amenant 2 acides aminés ensemble de façon à
ce qu'une enzyme puisse former un lien PEPTIDIQUE
entre les deux acides aminés adjacents.
L'ARNt suivant, avec son acide aminé, se lie au codon libre suivant
sur l'ARNm, amenant 2 acides aminés ensemble de façon à
ce qu'une enzyme puisse former un lien PEPTIDIQUE
entre les deux acides aminés adjacents.
![]() L'ARNm se déplace de nouveau d'un autre codon et le premier ARNt
vide est relâché.
L'ARNm se déplace de nouveau d'un autre codon et le premier ARNt
vide est relâché.
![]() etc.
etc.
Le processus continu tout le long de l'ARNm, la protéine grossissant au fur et à mesure, jusqu'au codon d'arrêt. Le ribosome est alors libéré et se re-sépare en sous-unités, à la recherche d'un autre codon de départ. En fait, les ribosomes font la queue devant les codons de départ et processent les protéines à la queuleuleu sur l'ARNm.
Un excellent site sur divers processus de la vie se trouve ici.
La régulation des gènes est un élément
essentiel pour le bon fonctionnement de tout organisme vivant. Par exemple,
E. coli contient ~4700 gènes, mais seulement ~1000 enzymes sont
requis dans le cytoplasme à un temps donné. D'autre part,
chaque cellule de notre corps contiennent tous nos gènes (~100,000)
mais n'en ont besoin que d'une partie dépendamment de leur localisation
et de leur condition à un temps donné.
Il apparaît évident que la régulation des gènes
prendra une importance toute particulière pour les organismes pluricellulaires:
personne n'aimerait voir un bras lui pousser dans le front, par exemple.
On peut aussi penser à des exemples plus dramatiques, comme les
difformités congénitales, le développement du cancer,
etc. Les cellules d'organismes pluricellulaires doivent donc développer
des moyens sophistiqués pour savoir où
et
quand
elles doivent exprimer tel ou tel gènes. Bien que le niveau de régulation
requis soit moins élevé, cela est aussi vrai pour les organismes
unicellulaires. Des "sens" doivent permettre de sonder les conditions environnementales
et des mécanismes de régulation
doivent
contrôler l'expression des gènes en conséquence. D'ailleurs
les premières études sur la régulation génétique
ont été faites sur des procaryotes. La compréhension
des ces mécanismes simples permettant de mieux comprendre par la
suite des schémas de contrôle plus complexes.
Avec le développement des réseaux d'aqueduc au début du siècle, E. coli s'est rapidement imposée comme un indicateur pratique de la pollution fécale de l'eau.
E.coli constitue plus de 80% des bactéries entériques.
La concentration en E. coli est proportionnelle à la concentration de plusieurs bactéries entériques pathogènes, mais se situe à un niveau beaucoup plus élevé.
Sa présence dans l'eau est indicatrice d'un risque de présence de pathogènes, son absence est une bonne garantie de l'absence de pathogènes entériques.
En général, elle est non-pathogène.
Elle est facile à identifier étant donné sa capacité peu commune à utiliser le lactose, un disaccharide composé de glucose et de galactose.
Un milieu permettant de l'identifier est basé sur cette caractéristique et a aussi pu servir à détecter les mutants.
De ce fait, la plupart des laboratoires sont équipés pour en faire la culture (simple).
Elle est donc connue, cultivée et étudiée par les microbiologistes depuis plus de 70 ans.
On pourrait dire qu'elle est devenue la bactérie la plus étudiée dans le monde.
Le métabolisme de dégradation du lactose chez E.coli a été largement étudié et est maintenant bien connu. Trois enzymes sont impliqués (voir ci-dessous).
L'enzyme responsable de séparer les deux hexoses est la ß-GALACTOSIDASE. Des tests colorimétriques simples, rapides, sensibles, reproductibles et peu dispendieux ont été développés permettant de mesurer l'activité de cette enzyme.
L'intérêt précoce qu'a connu E. coli
dans le domaine de la santé publique a fait en sorte qu'elle est
devenu le micro-organisme modèle sur lequel a porté une
part importante des recherches en génétique, en physiologie
et en biochimie microbienne. Cette concentration des efforts a entre autre
mené à la première mise au jour d'un mécanisme
de régulation génétique, la découverte
de l'opéron lactose par
Jacques Monod, Arthur B. Pardee et François Jacob au début
des années 60.

Figure 10. L'opéron lactose. Pour une discussion complète sur l'opéron lac, cliquez ici.
Monod , Pardee et Jacob découvrirent que le groupe d'enzymes responsables du métabolisme du lactose étaient INDUCTIBLES, c'est à dire que ces enzymes n'étaient synthétisées qu'en présence de lactose ou d'une substance analogue (tel le thiométhylgalactoside). De plus, si le lactose était éliminé du milieu, la synthèse de ces enzymes cessait en quelques secondes. Ce groupe de gènes, produits de façon coordonnée, fut appelé un OPÉRON (lactose, en l'occurence) et le lactose, un INDUCTEUR. L'opéron lactose est constitué des gènes de 3 enzymes: la ß-galactosidase (3072 pb) qui transforme le lactose en ses 2 hexoses constituants, la ß-galactoside permease (1251 pb), responsable du transport du lactose au travers la membrane plasmique et la thiogalactoside transacétylase (609 pb), une enzyme dont le rôle est encore inconnu.
Un grand nombre de mutants régulant la synthèse des enzymes du lactose ont été découverts entre 1940 et 1960. L'étude de ces mutants, a montré qu'un gène régulateur était responsable de la répression de la synthèse des enzymes du lactose et, qu'en fait, le lactose levait cette répression. Différents types de mutations affectaient le gène régulateur.
Type 1: la synthèse des enzymes du lactose ne s'arrêtait jamais. Leur expression était donc CONSTITUTIVE.
Type 2: les mutants devenaient non-inductibles, c'est à dire que le lactose n'induisait plus la synthèse des enzymes de l'opéron lac.
Type 3: même si le gène régulateur fonctionnait parfaitement bien, le système ne répondait plus au répresseur, même en absence de lactose. La synthèse des enzymes du lactose devenait donc là aussi constitutive.
Type 4: en absence de mutation notable du gène régulateur, les mutants ne synthétisaient pas les enzymes du lactose.
Avec l'accumulation des mutants et des résultats distincts, une compréhension globale du phénomène émergea:
![]() Le groupe de gènes des enzymes du lactose, l'OPÉRON
LACTOSE, se comportait comme une unité coordonnée:
produits toujours selon le même ratio.
Le groupe de gènes des enzymes du lactose, l'OPÉRON
LACTOSE, se comportait comme une unité coordonnée:
produits toujours selon le même ratio.
![]() Les cartes génétiques montrèrent que les gènes
contrôlant la synthèse des enzymes du lactose étaient
contigus
aux
gènes de ces enzymes.
Les cartes génétiques montrèrent que les gènes
contrôlant la synthèse des enzymes du lactose étaient
contigus
aux
gènes de ces enzymes.
![]() Ces cartes montrèrent aussi que le gène régulateur
était situé à une certaine distance des gènes
des enzymes du lactose, ou gènes de structure
(codant pour des polypeptides). Cela suggérait que le
produit du gène régulateur, le RÉPRESSEUR, devait
se déplacer à travers le cytoplasme jusqu'au site des gènes
des enzymes du lactose afin de réprimer leur synthèse. Cliquez
ici pour voir une protéine régulatrice se lier à
un site spécifique d'ADN.
Ces cartes montrèrent aussi que le gène régulateur
était situé à une certaine distance des gènes
des enzymes du lactose, ou gènes de structure
(codant pour des polypeptides). Cela suggérait que le
produit du gène régulateur, le RÉPRESSEUR, devait
se déplacer à travers le cytoplasme jusqu'au site des gènes
des enzymes du lactose afin de réprimer leur synthèse. Cliquez
ici pour voir une protéine régulatrice se lier à
un site spécifique d'ADN.
![]() L'explication qui mettait toutes ces constatations en relation était
que le gène régulateur produisait une PROTÉINE
RÉPRESSEUR se liant fortement à un site de l'ADN
proche du début de la transcription des gènes de l'opéron
lactose. Ce site d'adhésion sur l'ADN a été nommé
OPÉRATEUR.
En
se liant ainsi, le répresseur bloquait
le
mouvement de l'ARN polymerase sur les gènes de structures qui dirigeaient
la synthèse des protéines.
Cliquez
ici pour une autre description détaillée de l'opéron
lactose avec de très bonnes images.
L'explication qui mettait toutes ces constatations en relation était
que le gène régulateur produisait une PROTÉINE
RÉPRESSEUR se liant fortement à un site de l'ADN
proche du début de la transcription des gènes de l'opéron
lactose. Ce site d'adhésion sur l'ADN a été nommé
OPÉRATEUR.
En
se liant ainsi, le répresseur bloquait
le
mouvement de l'ARN polymerase sur les gènes de structures qui dirigeaient
la synthèse des protéines.
Cliquez
ici pour une autre description détaillée de l'opéron
lactose avec de très bonnes images.
![]() Il a donc été proposé qu'à son tour le lactose
agissait en se liant au répresseur et
en changeant sa forme fonctionnelle, causant ainsi son détachement
de l'opérateur, à la manière d'un effecteur allostérique.
Une fois le répresseur neutralisé, l'ARN polymérase
pouvait produire l'ARNm de ces gènes permettant ainsi la synthèse
des enzymes du lactose.
Il a donc été proposé qu'à son tour le lactose
agissait en se liant au répresseur et
en changeant sa forme fonctionnelle, causant ainsi son détachement
de l'opérateur, à la manière d'un effecteur allostérique.
Une fois le répresseur neutralisé, l'ARN polymérase
pouvait produire l'ARNm de ces gènes permettant ainsi la synthèse
des enzymes du lactose.
![]() Une mutation située sur le gène régulateur, affectant
le site de liaison à l'opérateur du répresseur, ou
encore une mutation de l'opérateur empêchant le répresseur
de s'y lier avait alors le même effet: empêcher le lien répresseur-opérateur
rendant ainsi l'expression des enzymes du lactose constitutive.
Une mutation située sur le gène régulateur, affectant
le site de liaison à l'opérateur du répresseur, ou
encore une mutation de l'opérateur empêchant le répresseur
de s'y lier avait alors le même effet: empêcher le lien répresseur-opérateur
rendant ainsi l'expression des enzymes du lactose constitutive.
![]() Inversement, si le gène régulateur mutait de façon
à modifier sur le répresseur le site de liaison du lactose
sans modifier le site de liaison à l'opérateur, le répresseur
ne quitterait jamais l'opérateur et la synthèse des enzymes
du lactose serait bloquée irréversiblement.
Inversement, si le gène régulateur mutait de façon
à modifier sur le répresseur le site de liaison du lactose
sans modifier le site de liaison à l'opérateur, le répresseur
ne quitterait jamais l'opérateur et la synthèse des enzymes
du lactose serait bloquée irréversiblement.
![]() Finalement, il a été proposé que l'ARN polymérase
devait se lier sur une séquence spécifique d'ADN située
au tout début de la série des gènes de structure.
Cette séquence fut nommée le PROMOTEUR.
Une mutation du promoteur faisant en sorte que l'ARN polymérase
ne puisse plus se lier empêcherait elle aussi de façon permanente
la synthèse des enzymes du lactose.
Finalement, il a été proposé que l'ARN polymérase
devait se lier sur une séquence spécifique d'ADN située
au tout début de la série des gènes de structure.
Cette séquence fut nommée le PROMOTEUR.
Une mutation du promoteur faisant en sorte que l'ARN polymérase
ne puisse plus se lier empêcherait elle aussi de façon permanente
la synthèse des enzymes du lactose.
Cette théorie fut par la suite entièrement vérifiée et sert maintenant de base pour comprendre la régulation génétique de tous les organismes, pour lesquels il existe cependant une multitude de variations sur un même thème.
Un exemple de ces variations est le cas des enzymes dont l'EXPRESSION est RÉPRESSIBLE. Ce sont des enzymes anaboliques qui ne sont pas synthétisées lorsque leur produit n'est pas nécessaire (ou en assez grande concentration). Ce schème de régulation est l'image miroir de la régulation des enzymes du lactose. Par exemple, les enzymes qui produisent les différents acides aminés ne sont nécessaires que lorsqu'il y a insuffisance d'approvisionnement externe. Cependant, dans le cas d'un approvisionnement abondant en acides aminés, il n'est plus nécessaire d'en produire. Le micro-organisme a donc besoin de pouvoir interrompre la synthèse des enzymes produisant ces acides aminés. Pour y arriver, il a, comme dans le cas précédent, un gène régulateur qui produit une protéine régulatrice, sauf que cette protéine n'a pas de site de liaison avec l'opérateur à moins qu'il n'y ai un excès en acides aminés. Dans le cas d'un excès, l'acide aminé en question se lie à un site allostérique sur la protéine régulatrice, ce qui modifie sa forme et augmente son affinité pour l'opérateur. Le répresseur se lie alors à l'opérateur et empêche la synthèse des enzymes structurales.